Name: Leopold
DNA-Replikation
Die DNA-Replikation beschreibt die exakte Verdopplung des Erbinformationsträgers, DNA. Die Verdopplung eines DNA-Stranges findet in der Regel vor einer Zellteilung, während der Synthese-Phase (S-Phase) der Mitose statt. Aus diesem Grund ist die Replikation stark mit der Zellteilung gekoppelt, da ohne ausreichende genetische Information sich die Zelle nicht teilen kann.
Die Vervielfältigung erfolgt nach einem semikonservativen Schema, was bedeutet, dass an den beiden zuvor getrennten Einzelsträngen des DNA-Doppelstrangs jeweils ein komplementärer Strang gebildet wird. Die Basenabfolge des neu gebildeten Strangs ist dabei bereits festgelegt, da jede Base eines DNA-Nukleotids nur mit einer festgelegten Base eine Verbindung eingehen kann (Adenin - Thymin ; Guanin - Cytosin) Die einzelnen Nukleotide verbinden sich mit ihrem komplementären Partner anhand der Wasserstoffbrückenbindungen.
1. Entspirialisierung und Öffnung des DNA-Doppelstrangs:
Das Enzym, Helicase, beginnt mit dem entwinden der DNA-Doppelhelix. Danach folgt die Trennung des Doppelstrangs welcher durch Wasserstoffbrückenbindungen zusammengehalten wird, diese jedoch keine hohe Bindungsenergie aufweisen und deshalb leicht durch einen enzymatischen Vorgang voneinander getrennt werden können. Durch die Auftrennung des Doppelstranges entstehen so am Startpunkt der Replikation zwei Replikationsgabeln, die während der Replikation entgegengestzt auseinanderlaufen. Damit sich die Basen nicht wieder über Wasserstoffbrückenbindungen paaren, halten sogenannte Einzelstrang-bindende Proteine (bei den Prokaryoten heißt dies SSB-Protein) die einzelnen Stränge auseinander.
2.Ergänzung der Stränge:
Im Anschluss der Öffnung folgt das Priming: An den nun freien Einzelsträngen wird durch eine RNA-Polymerase, die Primase, ein kurzes RNA-Stück, der Primer (etwa 10 Nukleotide) gesetzt. Die DNA-Polymerase benötigt den Primer als Starthilfe und hefetet an diesen DNA-Nucleotide an um den Tochterstrang synthetisieren zu können.
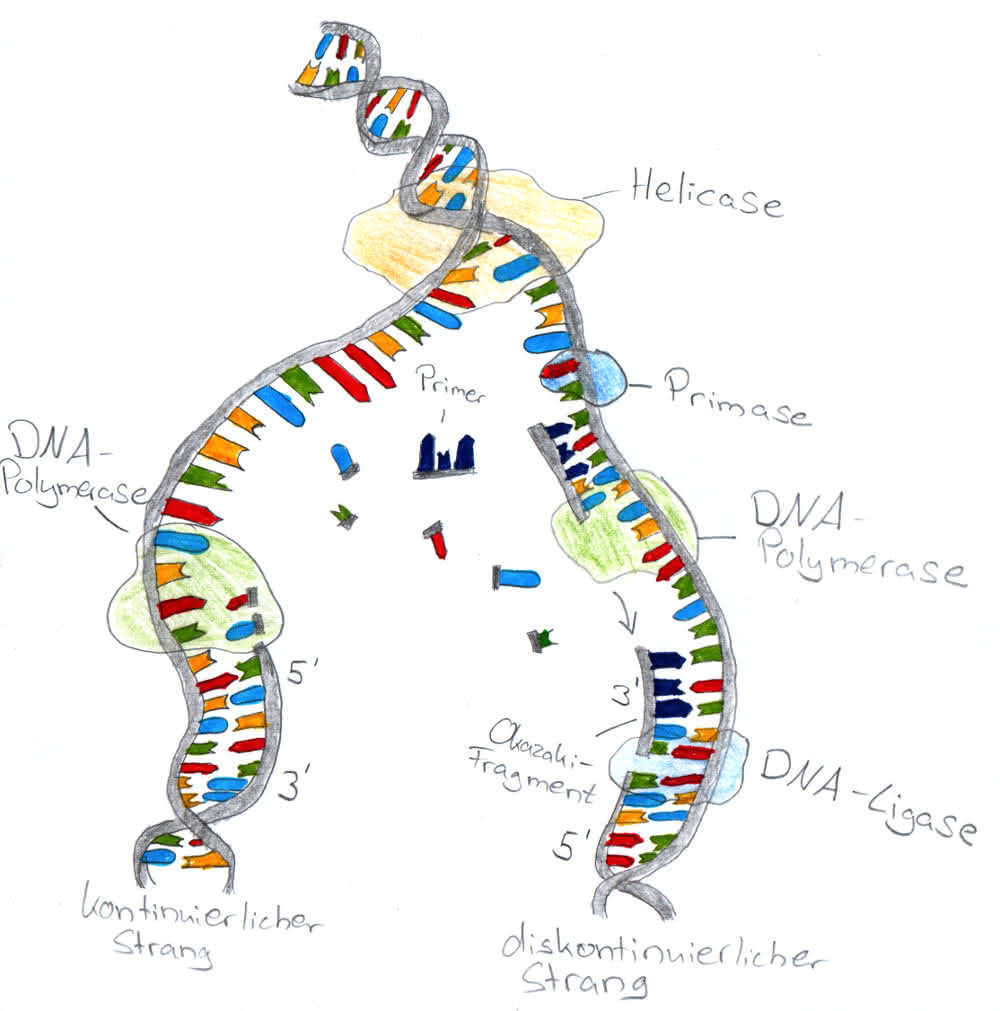
Beteiligte Enzyme: Ligase, verschiedene Polymerasen, Ligase, Primase, Helicase
Die beiden Stränge der DNA-Doppelhelix verlaufen gegenläufig. Der Elternstrang läuft in 3´zu 5´- Richtung, wo hingegen der andere in 5´- 3´Richtung verläuft. Aufgrund des festgelegten Verlaufs der DNA-Polymerase in 5´-3´- Richtung, verläuft die DNA-Polymerase lediglich am 3´-5´-Strang kontinuierlich, da die DNA-Polymerase der Verlaufsrichtung der Replikationsgabel folgt. Am anderen Strang verläuft die DNA-Polymerase diskontinuierlich, da sie dort nur von der Replikationsgabel weg verknüpfen kann. Die Synthetisierung bricht nach einem kurzen Stück ab und beginnt in der weiterschreitenden Gabel von neuem. Es entstehen dort kurze Sücke, neu synthetisierter DNA, welche nach ihrem Entdecker "Okazaki"- Stücke genannt werden. Für jedes dieser Stücke wurde vorher ein RNA-Primer synthetisiert. Mit dem Abbau der Okazaki-Stücke beginnt der Abbau des RNA-Primers, ebenfalls durch die DNA-Polymerase katalysiert. Die daraus entstandenen Lücken zwischen den vervollständigten Okazaki-Stücken werden von einem bestimmten Enzym , DNA-Ligase, zu einem durchgehenden Strang verbunden.
